Utilisation du format Parquet avec illustré à partir de quelques exemples
R



Ce tutoriel vise à offrir une approche complémentaire au guide d’utilisation des données du recensement au format Parquet publié sur https://ssphub.netlify.app pour accompagner la diffusion de celles-ci par l’Insee.
Il s’agit d’un tutoriel préparé pour l’atelier tuto@mate à l’EHESS le 5 novembre 2024. Ce tutoriel est exclusivement en R. Les slides de la présentation sont disponibles ci-dessous:
Pour retrouver une version Python équivalente, c’est ici.
Dérouler les slides ci-dessous ou cliquer ici pour afficher les slides en plein écran.
Il propose des exemples variés pour illustrer la simplicité d’usage du format Parquet. Parmi ceux-ci, à partir du recensement de la population:
- Recenser les Niçois qui ont emménagé depuis 2021 (Listing 1)
- Compter les Toulousains qui ont déménagé dans l’année (Listing 2)
- Pyramide des âges dans l’Aude, l’Hérault et la Haute-Garonne (Listing 3)
- Part des plus de 60 ans dans la population de chaque département (Listing 4)
- Part des résidences secondaires et des logements vacants dans le parc de chaque logement (Listing 5)
- Modes de transport pour chaque classe d’âge vivant dans Paris intra-muros (Listing 7)
A partir de la base permanent des équipements (BPE):
- Les établissements d’enseignement à Aubervilliers (Listing 8)
- Les établissements de sports à Saint Denis (Listing 10)
Librairies utilisées
Ce tutoriel utilisera plusieurs librairies R. Celles-ci peuvent être importées ainsi1
Téléchargement des fichiers
Pour commencer, nous allons télécharger les fichiers depuis internet pour limiter les échanges réseaux. Comme nous le verrons ultérieurement, ce n’est en fait pas indispensable car duckdb optimise les données téléchargées à chaque requête.
Voir le code pour télécharger les données
options(timeout = max(300, getOption("timeout")))
download_if_not_exists <- function(url, filename) {
if (!file.exists(filename)) {
download.file(url, filename)
message(paste("Downloaded:", filename))
} else {
message(paste("File already exists:", filename))
}
}
dir.create("data")
url_table_logement <- "https://static.data.gouv.fr/resources/recensement-de-la-population-fichiers-detail-logements-ordinaires-en-2020-1/20231023-123618/fd-logemt-2020.parquet"
url_table_individu <- "https://static.data.gouv.fr/resources/recensement-de-la-population-fichiers-detail-individus-localises-au-canton-ou-ville-2020-1/20231023-122841/fd-indcvi-2020.parquet"
url_doc_logement <- "https://www.data.gouv.fr/fr/datasets/r/c274705f-98db-4d9b-9674-578e04f03198"
url_doc_individu <- "https://www.data.gouv.fr/fr/datasets/r/1c6c6ab2-b766-41a4-90f0-043173d5e9d1"
# url_bpe <- "https://www.insee.fr/fr/statistiques/fichier/8217525/BPE23.parquet"
url_bpe <- "https://minio.lab.sspcloud.fr/lgaliana/diffusion/BPE23.parquet" #pb temporaire github actions avec insee.fr
url_bpe_metadata <- "https://www.insee.fr/fr/metadonnees/source/fichier/BPE23_table_passage.csv"
filename_table_logement <- "data/RPlogement.parquet"
filename_table_individu <- "data/RPindividus.parquet"
# Télécharge les fichiers
download_if_not_exists(url_table_logement, filename_table_logement)
download_if_not_exists(url_table_individu, filename_table_individu)
download_if_not_exists(url_doc_logement, "./data/dictionnaire_variables_logemt_2020.csv")
download_if_not_exists(url_doc_individu, "./data/dictionnaire_variables_indcvi_2020.csv")
download_if_not_exists(url_bpe, "./data/BPE2023.parquet")
download_if_not_exists(url_bpe_metadata, "./data/BPE2023_documentation.csv")- 1
- Ceci est nécessaire pour éviter une erreur si le téléchargement est un peu lent.
Nous aurons également besoin pour quelques illustrations d’un fond de carte des départements. Celui-ci peut être simplement récupéré grâce au package cartiflette2
Voir le code pour récupérer ce fond de carte
departements <- carti_download(
values="France",
crs=4326,
borders="DEPARTEMENT",
vectorfile_format="geojson",
filter_by="FRANCE_ENTIERE_DROM_RAPPROCHES",
source="EXPRESS-COG-CARTO-TERRITOIRE",
year=2022,
)Voir le code pour faire cette carte
ggplot(departements) +
geom_sf() +
theme_void()
Création de la base de données
En principe, duckdb fonctionne à la manière d’une base de données. Autrement dit, on définit une base de données et effectue des requêtes (SQL ou verbes tidyverse) dessus. Pour créer une base de données, il suffit de faire un read_parquet avec le chemin du fichier.
La base de données se crée tout simplement de la manière suivante:
Celle-ci peut être utilisée de plusieurs manières. En premier lieu, par le biais d’une requête SQL. dbGetQuery permet d’avoir le résultat sous forme de dataframe puisque la requête est déléguée à l’utilitaire duckdb qui est embarqué dans les fichiers de la librairie
out <- dbGetQuery(
con,
glue(
'SELECT * FROM read_parquet("{filename_table_individu}") LIMIT 5'
)
)
outLa chaîne d’exécution ressemble ainsi à celle-ci:

Même si DuckDB simplifie l’utilisation du SQL en proposant de nombreux verbes auxquels on est familier en R ou Python, SQL n’est néanmoins pas toujours le langage le plus pratique pour chaîner des opérations nombreuses. Pour ce type de besoin, le tidyverse offre une grammaire riche et cohérente. Il est tout à fait possible d’interfacer une base duckdb au tidyverse. On pourra donc utiliser nos verbes préférés (mutate, filter, etc.) sur un objet duckdb: une phase préliminaire de traduction en SQL sera automatiquement mise en oeuvre:
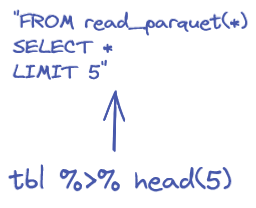
L’équivalent tidyverse de la requête précédente est la fonction head
Le fait de passer par l’intermédiaire de duckdb et un fichier Parquet permet d’optimiser les besoins mémoire de R. En effet, il n’est pas nécessaire d’ouvrir un fichier dans son ensemble, le transformer en objet R pour n’utiliser qu’une partie des données. Nous verrons ultérieurement la manière dont les besoins mémoires sont minimisés grâce au combo duckdb & Parquet.
Il est à noter que l’objet renvoyé par R n’est pas, à ce stade, un dataframe classique. Il s’agit d’un dataframe lazy, en attente d’exécution complète:
[1] "tbl_duckdb_connection" "tbl_dbi" "tbl_sql"
[4] "tbl_lazy" "tbl" Pour exécuter sur l’ensemble de la base, il faut faire un collect. Ceci déclenchera l’ensemble de la chaîne d’opérations préparée avec duckdb. On peut voir les opérations antérieures comme un plan, qui attend d’être mis en oeuvre.
collect déclenche les calculs. Pour bénéficier des avantages de l’exécution déportée sur duckdb, il vaut mieux préparer bien son plan d’exécution. Cela signifie que les opérations gourmandes en calcul doivent être, dans la mesure du possible, avant le collect.
Par exemple, faire
est une mauvaise pratique. Cela ramène trop de données inutiles dans R, ce qui risque de provoquer des problèmes de mémoire. Si on ne veut que les premières lignes de notre jeu de données, il faut faire
Cette fois, l’échantillonnage se fait avant l’exécution par duckdb.
Enfin, nous pouvons importer les dictionnaires des variables qui pourront nous servir ultérieurement:
Ouvrir un fichier Parquet
Requêtes sur les colonnes (SELECT)
L’une des forces du format Parquet est de simplifier l’import de fichiers volumineux qui ne comportent que quelques colonnes nous intéressant. Par exemple, la table des individus comporte 88 colonnes, il est peu probable qu’une seule analyse s’intéresse à toutes celles-ci (ou elle risque d’être fort indigeste).
Comme cela est illustré dans Tip 1, la différence de volumétrie entre un fichier non filtré et un fichier filtré est importante.
glue(
"SELECT IPONDI AS poids, AGED, VOIT ",
"FROM read_parquet(\"{filename_table_individu}\") "
)SELECT IPONDI AS poids, AGED, VOIT FROM read_parquet("data/RPindividus.parquet") query <- glue(
"SELECT IPONDI AS poids, AGED, VOIT ",
"FROM read_parquet(\"{filename_table_individu}\") "
)
dbGetQuery(
con,
query %>% head(10)
)Parquet et DuckDB
Pour réduire la volumétrie des données importées, il est possible de mettre en oeuvre deux stratégies:
- N’importer qu’un nombre limité de colonnes
- N’importer qu’un nombre limité de lignes
Comme cela a été évoqué dans les slides, le format Parquet est particulièrement optimisé pour le premier besoin. C’est donc généralement la première optimisation mise en oeuvre. Pour s’en convaincre on peut regarder la taille des données importées dans deux cas:
- On utilise beaucoup de lignes mais peu de colonnes
- On utilise beaucoup de colonnes mais peu de lignes
Pour cela, nous utilisons la fonction SQL EXPLAIN ANALYZE disponible dans duckdb. Elle décompose le plan d’exécution de duckdb, ce qui nous permettra de comprendre la stratégie d’optimisation. Elle permet aussi de connaître le volume de données importées lorsqu’on récupère un fichier d’internet. En effet, duckdb est malin: plutôt que de télécharger un fichier entier pour n’en lire qu’une partie, la librairie est capable de n’importer que les blocs du fichier qui l’intéresse.
Ceci nécessite l’utilisation de l’extension httpfs (un peu l’équivalent des library de R en duckdb). Elle s’installe et s’utilise de la manière suivante
Demandons à DuckDB d’exécuter la requête “beaucoup de colonnes, pas beaucoup de lignes” et regardons le plan d’exécution et les informations données par DuckDB:
Voir le plan : “beaucoup de colonnes, pas beaucoup de lignes”
glue(
'EXPLAIN ANALYZE ',
'SELECT * FROM read_parquet("{url_bpe}") LIMIT 5'
)EXPLAIN ANALYZE SELECT * FROM read_parquet("https://minio.lab.sspcloud.fr/lgaliana/diffusion/BPE23.parquet") LIMIT 5plan <- dbGetQuery(
con,
glue(
'EXPLAIN ANALYZE ',
'SELECT * FROM read_parquet("{url_bpe}") LIMIT 5'
)
)print(plan)analyzed_plan
┌─────────────────────────────────────┐
│┌───────────────────────────────────┐│
││ Query Profiling Information ││
│└───────────────────────────────────┘│
└─────────────────────────────────────┘
EXPLAIN ANALYZE SELECT * FROM read_parquet("https://minio.lab.sspcloud.fr/lgaliana/diffusion/BPE23.parquet") LIMIT 5
┌─────────────────────────────────────┐
│┌───────────────────────────────────┐│
││ HTTP Stats: ││
││ ││
││ in: 164.5 MiB ││
││ out: 0 bytes ││
││ #HEAD: 1 ││
││ #GET: 5 ││
││ #PUT: 0 ││
││ #POST: 0 ││
│└───────────────────────────────────┘│
└─────────────────────────────────────┘
┌─────────────────────────────────────┐
│┌───────────────────────────────────┐│
││ Total Time: 16.70s ││
│└───────────────────────────────────┘│
└─────────────────────────────────────┘
┌───────────────────────────┐
│ RESULT_COLLECTOR │
│ ─ ─ ─ ─ ─ ─ ─ ─ ─ ─ ─ │
│ 0 │
│ (0.00s) │
└─────────────┬─────────────┘
┌─────────────┴─────────────┐
│ EXPLAIN_ANALYZE │
│ ─ ─ ─ ─ ─ ─ ─ ─ ─ ─ ─ │
│ 0 │
│ (0.00s) │
└─────────────┬─────────────┘
┌─────────────┴─────────────┐
│ LIMIT │
│ ─ ─ ─ ─ ─ ─ ─ ─ ─ ─ ─ │
│ 5 │
│ (0.00s) │
└─────────────┬─────────────┘
┌─────────────┴─────────────┐
│ READ_PARQUET │
│ ─ ─ ─ ─ ─ ─ ─ ─ ─ ─ ─ │
│ AN │
│ NOMRS │
│ CNOMRS │
│ NUMVOIE │
│ INDREP │
│ TYPVOIE │
│ LIBVOIE │
│ CADR │
│ CODPOS │
│ DEPCOM │
│ DEP │
│ REG │
│ DOM │
│ SDOM │
│ TYPEQU │
│ SIRET │
│ STATUT_DIFFUSION │
│ CANTINE │
│ INTERNAT │
│ RPI │
│ EP │
│ CL_PGE │
│ SECT │
│ ACCES_AIRE_PRATIQUE │
│ ACCES_LIBRE │
│ ACCES_SANITAIRE │
│ ACCES_VESTIAIRE │
│ CAPACITE_D_ACCUEIL │
│ PRES_DOUCHE │
└───────────────────────────┘ Voir le plan : “peu de colonnes, beaucoup de lignes”
plan <- dbGetQuery(
con,
glue(
'EXPLAIN ANALYZE ',
'SELECT TYPEQU, LONGITUDE, LATITUDE FROM read_parquet("{url_bpe}") LIMIT 10000'
)
)print(plan)analyzed_plan
┌─────────────────────────────────────┐
│┌───────────────────────────────────┐│
││ Query Profiling Information ││
│└───────────────────────────────────┘│
└─────────────────────────────────────┘
EXPLAIN ANALYZE SELECT TYPEQU, LONGITUDE, LATITUDE FROM read_parquet("https://minio.lab.sspcloud.fr/lgaliana/diffusion/BPE23.parquet") LIMIT 10000
┌─────────────────────────────────────┐
│┌───────────────────────────────────┐│
││ HTTP Stats: ││
││ ││
││ in: 38.5 MiB ││
││ out: 0 bytes ││
││ #HEAD: 1 ││
││ #GET: 8 ││
││ #PUT: 0 ││
││ #POST: 0 ││
│└───────────────────────────────────┘│
└─────────────────────────────────────┘
┌─────────────────────────────────────┐
│┌───────────────────────────────────┐│
││ Total Time: 8.28s ││
│└───────────────────────────────────┘│
└─────────────────────────────────────┘
┌───────────────────────────┐
│ RESULT_COLLECTOR │
│ ─ ─ ─ ─ ─ ─ ─ ─ ─ ─ ─ │
│ 0 │
│ (0.00s) │
└─────────────┬─────────────┘
┌─────────────┴─────────────┐
│ EXPLAIN_ANALYZE │
│ ─ ─ ─ ─ ─ ─ ─ ─ ─ ─ ─ │
│ 0 │
│ (0.00s) │
└─────────────┬─────────────┘
┌─────────────┴─────────────┐
│ LIMIT │
│ ─ ─ ─ ─ ─ ─ ─ ─ ─ ─ ─ │
│ 10000 │
│ (0.00s) │
└─────────────┬─────────────┘
┌─────────────┴─────────────┐
│ READ_PARQUET │
│ ─ ─ ─ ─ ─ ─ ─ ─ ─ ─ ─ │
│ TYPEQU │
│ LONGITUDE │
│ LATITUDE │
│ ─ ─ ─ ─ ─ ─ ─ ─ ─ ─ ─ │
│ EC: 2773420 │
│ ─ ─ ─ ─ ─ ─ ─ ─ ─ ─ ─ │
│ 30720 │
│ (11.14s) │
└───────────────────────────┘ La comparaison de ces plans d’exécution montre l’intérêt de faire un filtre sur les colonnes : les besoins computationnels sont drastiquement diminués. Le filtre sur les lignes n’arrive que dans un second temps, une fois les colonnes sélectionnées.
Pourquoi seulement un rapport de 1 à 4 entre le poids des deux fichiers ? C’est parce que nos requêtes comportent toute deux la variable IPONDI (les poids à utiliser pour extrapoler l’échantillon à la population) qui est à haute précision là où beaucoup d’autres colonnes comportent un nombre réduit de modalités et sont donc peu volumineuses.
DuckDB propose également des fonctionnalités pour extraire des colonnes à travers des expressions régulières. Cette approche est également possible avec le tidyverse
Requêtes sur les lignes (WHERE)
Les filtres sur les observations peuvent être faits à partir de critères sur plusieurs colonnes. Par exemple, pour ne conserver que les observations de la ville de Nice où la date d’emménagement est postérieure à 2020, la requête suivante peut être utilisée :
dbGetQuery(
con,
glue(
"FROM read_parquet(\"{filename_table_logement}\") ",
"SELECT * ",
"WHERE COMMUNE = '06088' and AEMM > 2020 ",
"LIMIT 10"
)
)Statistiques agrégées
Exemples sans groupes
La fonction DISTINCT appliquée à la variable ARM permet d’extraire la liste des codes arrondissements présents dans la base de données.
query <- glue_sql(
"FROM read_parquet({filename_table_logement}) ",
"SELECT DISTINCT(ARM) ",
"WHERE NOT CONTAINS(ARM, 'ZZZZZ') ",
"ORDER BY ARM",
.con=con
)
paste(dbGetQuery(con, query)$ARM, collapse = ", ")[1] "13201, 13202, 13203, 13204, 13205, 13206, 13207, 13208, 13209, 13210, 13211, 13212, 13213, 13214, 13215, 13216, 69381, 69382, 69383, 69384, 69385, 69386, 69387, 69388, 69389, 75101, 75102, 75103, 75104, 75105, 75106, 75107, 75108, 75109, 75110, 75111, 75112, 75113, 75114, 75115, 75116, 75117, 75118, 75119, 75120"Il est possible d’extraire des statistiques beaucoup plus raffinées par le biais d’une requête SQL plus complexe. Par exemple pour calculer le nombre d’habitants de Toulouse qui ont changé de logement en un an:
Warning: Missing values are always removed in SQL aggregation functions.
Use `na.rm = TRUE` to silence this warning
This warning is displayed once every 8 hours.query <- glue(
"FROM read_parquet(\"{filename_table_logement}\") ",
"SELECT CAST(SUM(IPONDL*CAST(INPER AS INT)) AS INT) ",
"AS habitants_toulouse_demenagement ",
"WHERE COMMUNE == '31555' AND IRANM NOT IN ('1', 'Z') AND INPER != 'Y'"
)
dbGetQuery(con, query)Statistiques par groupe
SQL et dplyr permettent d’aller loin dans la finesse des statistiques descriptives mises en oeuvre. Cela sera illustré à l’aide de plusieurs exemples réflétant des statistiques pouvant être construites grâce à ces données détaillées.
Exemple 1: pyramide des âges dans l’Aude, l’Hérault et le Gard
Le premier exemple est un comptage sur trois départements. Il illustre la démarche suivante:
- On se restreint aux observations d’intérêt (ici 3 départements)
- On applique la fonction
summarisepour calculer une statistique par groupe, en l’occurrence la somme des pondérations - On retravaille les données
Ensuite, une fois que nos données sont récupérées dans R, on peut faire la figure avec ggplot
pyramide_ages <- table_individu %>%
filter(DEPT %in% c('11', '31', '34')) %>%
group_by(AGED, departement = DEPT) %>%
summarise(individus = sum(IPONDI), .groups = "drop") %>%
arrange(departement, AGED) %>%
collect()
ggplot(pyramide_ages, aes(x = AGED, y = individus)) +
geom_bar(aes(fill = departement), stat = "identity") +
geom_vline(xintercept = 18, color = "grey", linetype = "dashed") +
facet_wrap(~departement, scales = "free_y", nrow = 3) +
theme_minimal() +
labs(y = "Individus recensés", x = "Âge")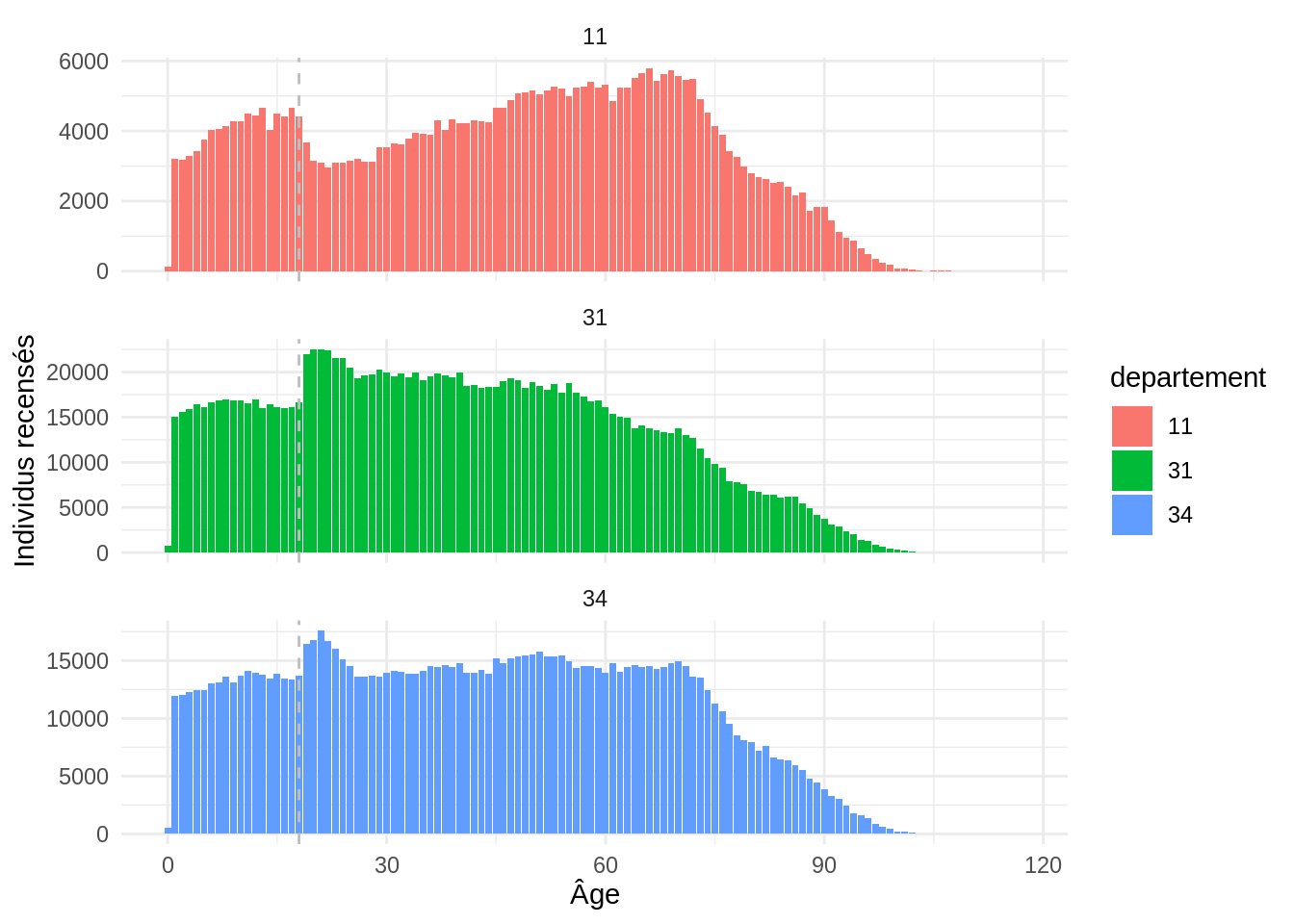
Exemple 2: répartition des plus de 60 ans par département
L’objectif de ce deuxième exemple est d’illustrer la construction d’une statistique un peu plus complexe et la manière de projeter celle-ci sur une carte.
Pour avoir la répartition des plus de 60 ans par département, quelques lignes de dplyr suffisent:
part_population_60_plus <- table_individu %>%
group_by(DEPT) %>%
summarise(
total_population = sum(IPONDI), # Population totale
population_60_plus = sum(IPONDI[AGED > 60]) # Population de plus de 60 ans
) %>%
mutate(pourcentage_60_plus = population_60_plus / total_population * 100) %>%
collect()
part_population_60_plusIl ne reste plus qu’à projeter ceci sur une carte. Pour cela, un join à notre fond de carte suffit. Comme les données sont agrégées et déjà dans R, il n’y a rien de spécifique à duckdb ici.
Association de part_population_60_plus au fond de carte des départements
# Joindre les données au fond de carte des départements
departements_60_plus_sf <- departements %>%
inner_join(
part_population_60_plus,
by = c("INSEE_DEP" = "DEPT")
)Finalement, il ne reste plus qu’à produire la carte:
ggplot(departements_60_plus_sf) +
geom_sf(aes(fill = pourcentage_60_plus)) +
scale_fill_fermenter(n.breaks = 5, palette = "PuBuGn", direction = 1) +
theme_void() +
labs(
title = "Part des personnes de plus de 60 ans par département",
caption = "Source: Insee, Fichiers détails du recensement de la population",
fill = "Part (%)"
)
Si on préfère représenter ceci sous forme de tableau, on peut utiliser le package gt.
Code pour avoir le classement des départements pour lesquels la population de plus de 60 ans est la plus importante
top_population <- part_population_60_plus %>%
left_join(
departements %>% select(INSEE_DEP, LIBELLE_DEPARTEMENT ) %>% st_set_geometry(NULL),
by = c("DEPT" = "INSEE_DEP")
) %>%
mutate(departement = paste0(LIBELLE_DEPARTEMENT, " (", DEPT , ")")) %>%
select(-DEPT, -LIBELLE_DEPARTEMENT) %>%
arrange(desc(pourcentage_60_plus)) %>%
select(DEPT = departement, everything()) %>%
head(10)
gt(
top_population
) %>%
gt_plt_bar_pct(
column = pourcentage_60_plus,
scaled = TRUE,
labels = TRUE
) %>%
fmt_number(
columns = c("total_population", "population_60_plus"),
decimals = 0,
sep_mark = " "
) %>%
fmt_number(
columns = c("pourcentage_60_plus"),
decimals = 1
) %>%
cols_label(
DEPT = md("**Département**"),
total_population = md("**Population**"),
population_60_plus = md("**Population de plus de 60 ans**"),
pourcentage_60_plus = md("*Part (%)*")
)| Département | Population | Population de plus de 60 ans | Part (%) |
|---|---|---|---|
| Creuse (23) | 116 178 | 45 561 |
39.2%
|
| Lot (46) | 174 397 | 67 122 |
38.5%
|
| Nièvre (58) | 202 728 | 76 108 |
37.5%
|
| Dordogne (24) | 413 180 | 153 405 |
37.1%
|
| Cantal (15) | 144 321 | 51 594 |
35.7%
|
| Gers (32) | 191 737 | 68 261 |
35.6%
|
| Indre (36) | 218 469 | 77 537 |
35.5%
|
| Charente-Maritime (17) | 655 648 | 232 006 |
35.4%
|
| Allier (03) | 335 380 | 118 481 |
35.3%
|
| Corrèze (19) | 239 286 | 84 511 |
35.3%
|
Exemple 3: part des résidences secondaires et des logements vacants
Il est tout à fait possible de faire des étapes antérieures de préparation de données, notamment de création de variables avec mutate.
L’exemple suivant illustre la préparation de données avant la construction de statistiques descriptives de la manière suivante:
- Création d’une variable de département à partir du code commune
- Décompte des logements par département
`summarise()` has grouped output by "DEPT". You can override using the
`.groups` argument.# Jointure avec le fond de carte des départements
parc_locatif_sf <- departements %>%
inner_join(
parc_locatif,
by = c("INSEE_DEP" = "DEPT"),
relationship = "many-to-many"
) %>%
group_by(INSEE_DEP) %>%
mutate(p = n/sum(n)) %>%
ungroup- 1
-
On a des clés dupliquées dans le fond
cartiflette(le zoom pour l’Ile de France) et dans le dataframe (4 valeurs par dep)
Code pour produire la carte
# Carte: Part des résidences secondaires
carte1 <- ggplot(parc_locatif_sf %>% filter(CATL == "3")) +
geom_sf(aes(fill = p), color = "white") +
scale_fill_fermenter(
n.breaks = 5,
palette = "RdPu",
direction = 1,
labels = scales::label_percent(
scale_cut = scales::cut_short_scale()
)
) +
theme_void() +
labs(
fill = "Part dans le\nparc de logement (%)",
title = "Cartographie des résidences secondaires",
caption = "Source: Insee, Fichiers détails du recensement de la population"
)
# Carte: Part des logements vacants
carte2 <- ggplot(parc_locatif_sf %>% filter(CATL == "4")) +
geom_sf(aes(fill = p), color = "white") +
scale_fill_fermenter(
n.breaks = 5,
palette = "RdPu",
direction = 1,
labels = scales::label_percent(
scale_cut = scales::cut_short_scale()
)
) +
theme_void() +
labs(
fill = "Part dans le\nparc de logement (%)",
title = "Cartographie des logements vacants",
caption = "Source: Insee, Fichiers détails du recensement de la population"
)
carte1
carte2
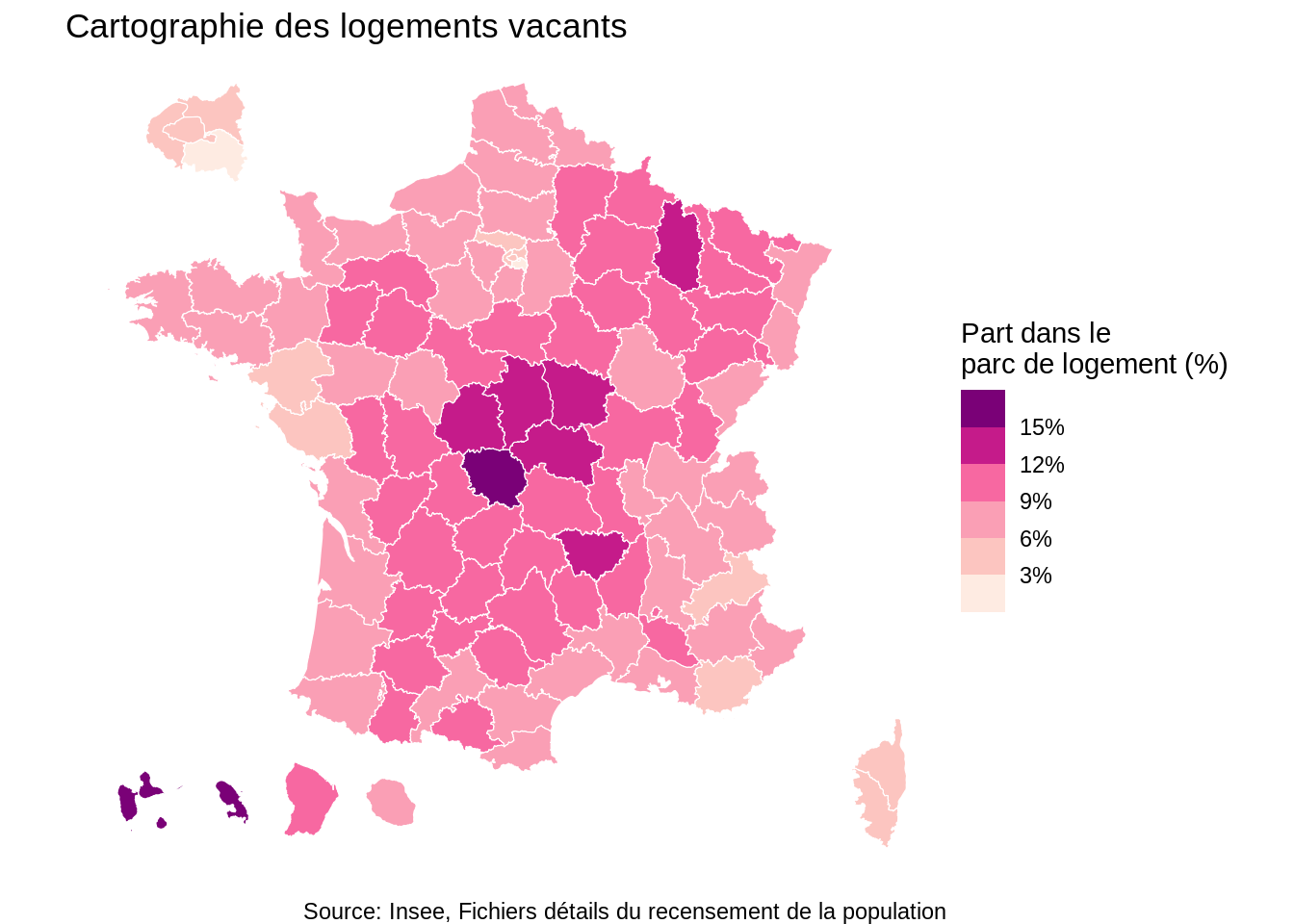
Enrichissement grâce aux jointures
Un autre cas d’usage classique est l’association de sources de données pour enrichir celles-ci à partir d’informations communes à partir de dimensions communes.
Pour illustrer cette approche, nous allons montrer comment faire ceci en associant notre source de données aux modalités de la variable sur le mode de transport issue de la documentation de notre source. Cela nous permet d’enrichir notre jeu de données d’informations intéressantes pour la représentation graphique et l’analyse.
`summarise()` has grouped output by "DEPT" and "AGEREVQ". You can override
using the `.groups` argument.# Part des modes de transport par âge et département
transports_age <- transports_age %>%
group_by(DEPT, AGEREVQ) %>%
filter(DEPT == 75) %>%
mutate(p = n/sum(n))
# Ajout du libellé du mode de transport
transports_age <- transports_age %>%
inner_join(
y = documentation_individus %>% filter(COD_VAR == "TRANS"),
by = c("TRANS" = "COD_MOD")
)
# Graphique
ggplot(transports_age, aes(x = as.numeric(AGEREVQ), y = p, color = factor(LIB_MOD))) +
geom_line() +
geom_point(shape = 17) +
scale_x_continuous(limits = c(20,70)) +
labs(color = "", x = "Age", y = "Proportion de la classe d'âge\nutilisant ce moyen") +
scale_y_continuous(labels = scales::percent) +
theme_minimal() +
theme(legend.position = "bottom") +
guides(color=guide_legend(nrow=3,byrow=TRUE))Warning: Removed 40 rows containing missing values or values outside the scale range
(`geom_line()`).Warning: Removed 40 rows containing missing values or values outside the scale range
(`geom_point()`).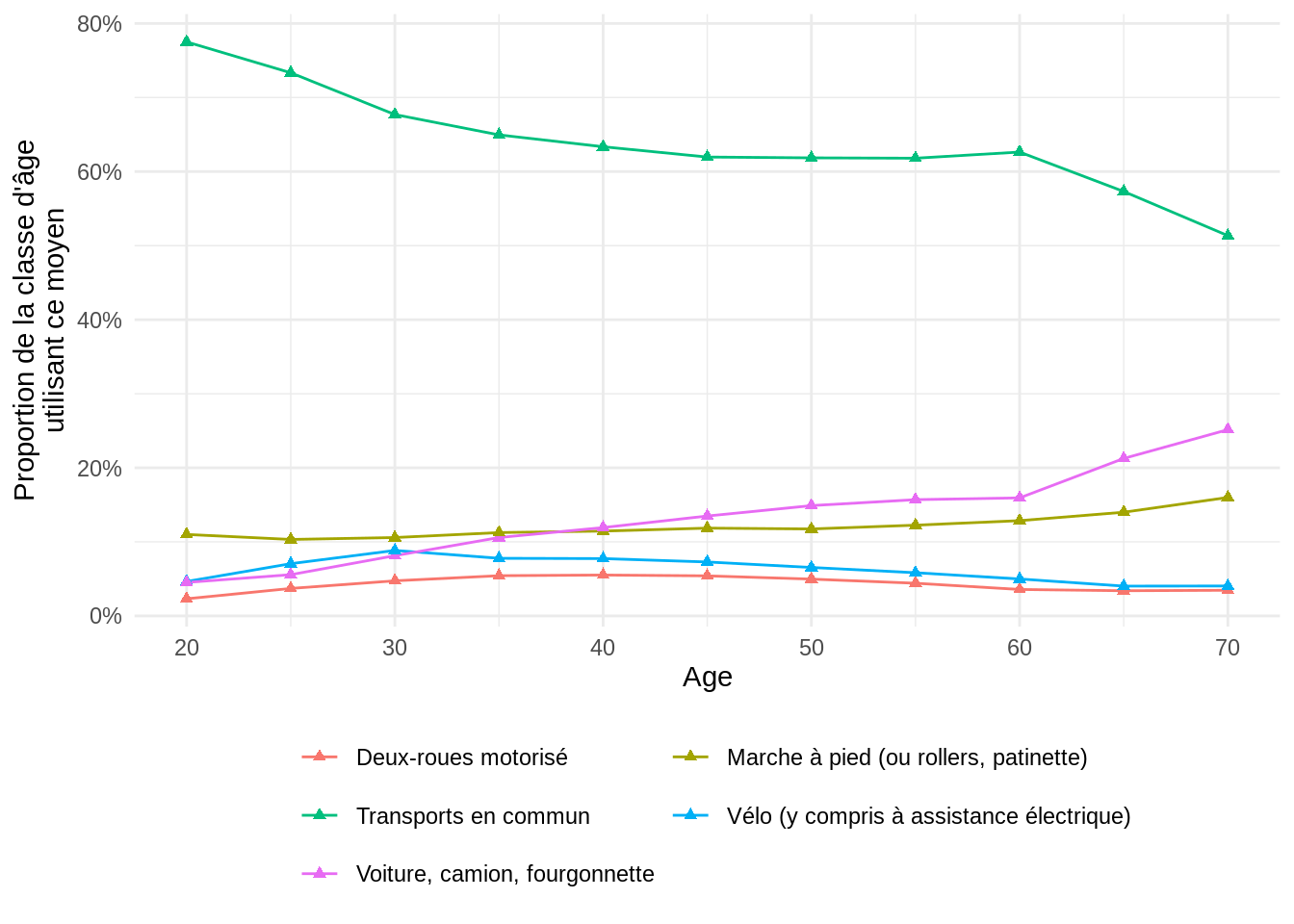
Le format Parquet pour les données géographiques
Pour illustrer l’intérêt de la diffusion de données géographiques, nous allons utiliser la base permanente des équipements, une source qui recense les équipements publics sur le territoire. Le millésime 2023 est, pour la première fois, diffusé au format Parquet. Le dictionnaire des variables de cette source est disponible ici.
GeoParquet
Il existe une extension du format Parquet pour les données géographiques: le GeoParquet. Ce format de fichier permet de gérer nativement les opérations sur les bounding box des géométries de chaque observation. Cela permet, par exemple, de filtrer les géométries appartenant à un certain rayon. Pour cela, il faut installer l’extension SPATIAL de duckdb.
Lorsque la géométrie des données se restreint à des coordonnées ponctuelles, c’est-à-dire à un couple longitude-latitude, le format GeoParquet n’apporte pas de gain particulier. Un Parquet classique, avec les longitudes et latitudes dans deux colonnes, suffit. C’est pour cette raison que la BPE est diffusée sous la forme classique d’un Parquet et non d’un GeoParquet.
[1] TRUE[1] TRUENous allons mettre en oeuvre une série d’opérations pour montrer que ce fichier fonctionne de manière identique à précédemment.
- En premier lieu, pour illustrer que ce fichier fonctionne de manière identique à précédemment, faisons un filtre à partir des variables de cette base (les géomaticiens parleraient de filtre attributaire). Récupérons exclusivement les établissements d’enseignement dans Aubervilliers3. On crée une variable temporaire
parquet_datadans SQL. - On joint, directement depuis SQL, au fichier de documentation.
La requête est un petit peu plus complexe que précédemment, la voici:
query <- glue(
"WITH parquet_data AS (",
"FROM read_parquet('./data/BPE2023.parquet')",
"SELECT * ",
"WHERE DEPCOM = '93001'",
" AND starts_with(TYPEQU, 'C')",
" AND NOT (starts_with(TYPEQU, 'C6') OR starts_with(TYPEQU, 'C7'))",
")",
"SELECT parquet_data.*, csv_data.Libelle_TYPEQU",
"FROM parquet_data",
"JOIN read_csv_auto('data/BPE2023_documentation.csv') AS csv_data",
" ON parquet_data.TYPEQU = csv_data.TYPEQU",
.sep = "\n"
)
queryWITH parquet_data AS (
FROM read_parquet('./data/BPE2023.parquet')
SELECT *
WHERE DEPCOM = '93001'
AND starts_with(TYPEQU, 'C')
AND NOT (starts_with(TYPEQU, 'C6') OR starts_with(TYPEQU, 'C7'))
)
SELECT parquet_data.*, csv_data.Libelle_TYPEQU
FROM parquet_data
JOIN read_csv_auto('data/BPE2023_documentation.csv') AS csv_data
ON parquet_data.TYPEQU = csv_data.TYPEQUOn peut directement l’exécuter avec duckdb et obtenir le dataframe suivant:
etab_enseignement_auber <- dbGetQuery(con, query)
head(etab_enseignement_auber)nrow(etab_enseignement_auber)[1] 61Nous avons 61 établissements d’enseignement recensés dans Aubervilliers. Transformons en dataframe géographique pour pouvoir les représenter sur une carte:
etab_enseignement_auberleaflet(etab_enseignement_auber) %>%
addTiles() %>%
addMarkers(popup = ~paste0(
"<b>", NOMRS, "</b><br>",
"<b>Type d'établissement</b>: ", tolower(Libelle_TYPEQU), " (", TYPEQU, ")")
) %>%
fitBounds(bounds[1], bounds[2], bounds[3], bounds[4])Pour ce fichier, la mise en oeuvre de filtres géographiques revient à faire un filtre classique.
bpe2023metadata_bpe <- readr::read_csv2(
"./data/BPE2023_documentation.csv"
)ℹ Using "','" as decimal and "'.'" as grouping mark. Use `read_delim()` for more control.Rows: 209 Columns: 6
── Column specification ────────────────────────────────────────────────────────
Delimiter: ";"
chr (6): TYPEQU, Libelle_TYPEQU, SDOM, Libelle_SDOM, DOM, Libelle_DOM
ℹ Use `spec()` to retrieve the full column specification for this data.
ℹ Specify the column types or set `show_col_types = FALSE` to quiet this message.equipements_sportifs_saint_denis <- bpe2023 %>%
filter(
starts_with(TYPEQU, "F1"),
between(LONGITUDE, 2.327943, 2.399054),
between(LATITUDE, 48.912967, 48.939077)
) %>%
collect() %>%
left_join(metadata_bpe, by = "TYPEQU")- La magie de
DuckDBen action ! La fonctionstarts_withvient du packageduckdb(cf. Tip 2)
starts_with ?
Quand dbplyr ne connait pas une fonction, il la passe telle qu’elle à duckdb. Ce dernier package va alors essayer d’utiliser une fonction ayant ce nom dans duckdb. En l’occurrence, starts_with existe dans duckdb, cette fonction sera donc utilisée et on n’aura pas d’erreur ?
Pourquoi faire ceci plutôt qu’utiliser une fonction pré-implémentée de stringr ? Car les calculs sont faits hors de R, directement par duckdb. Utiliser une fonction R impliquerait de changer l’ordre d’exécution de nos requêtes pour faire le collect avant le filter.
query <- glue(
"WITH parquet_data AS (",
" FROM read_parquet('./data/BPE2023.parquet')",
" SELECT *",
" WHERE",
" starts_with(TYPEQU, 'F1')",
" AND longitude BETWEEN 2.327943 AND 2.399054",
" AND latitude BETWEEN 48.912967 AND 48.939077",
")",
"SELECT parquet_data.*, csv_data.Libelle_TYPEQU",
"FROM parquet_data",
"JOIN read_csv_auto('https://www.insee.fr/fr/metadonnees/source/fichier/BPE23_table_passage.csv') AS csv_data",
"ON parquet_data.TYPEQU = csv_data.TYPEQU",
.sep = "\n"
)equipements_sportifs_saint_denis <- dbGetQuery(con, query)
head(equipements_sportifs_saint_denis)icons <- awesomeIconList(
running = makeAwesomeIcon(text = fa("running"), markerColor = "red")
)
leaflet(equipements_sportifs_saint_denis) %>%
addTiles() %>%
addAwesomeMarkers(
icon=icons['running'],
popup = ~paste0(
"<b>", NOMRS, "</b><br>",
"<b>Type d'établissement</b>: ", tolower(Libelle_TYPEQU), " (", TYPEQU, ")")
) %>%
fitBounds(bounds[1], bounds[2], bounds[3], bounds[4])Pour aller plus loin sur les filtres géographiques mis en oeuvre, il serait pratique d’utiliser l’extension spatiale qui implémente de nombreuses fonctions de manipulation de données spatiales. Nous laisserons les curieux creuser.
Conclusion
Ce tutoriel a permis à partir de quelques exemples de comprendre l’intérêt du format Parquet pour l’exploitation de données structurées. Les principaux enseignements à garder en mémoire sont les suivants:
-
duckdbest un écosystème qui rend très simple l’utilisation de ce type de fichiers. Que ce soit par le biais de la librairieduckdbou par l’intermédiaire dutidyverse, on bénéficie des avantages du monde de la base de données sans ses inconvénients. -
duckdbest très performant en lecture mais pour tirer pleinement parti du formatParquet, il est utile de faire très tôt les filtres sur les lignes et les colonnes. La lecture d’un fichier volumineux (5Go en CSV) devient presque instantanée grâce au comboParquet&duckdb.
Ce tutoriel était une introduction à l’utilisation du format Parquet. Il n’évoque pas, à l’heure actuelle, deux dimensions intéressantes de ce format :
- La possibilité d’avoir des
Parquetpartitionné qui permettent d’accélérer les requêtes ne s’appuyant sur un sous-ensemble de lignes - Les excellentes performances de
Parquetavec un système de stockage cloud typeS3(technologie derrière leSSPCloud)
Footnotes
Si vous avez clôné le dépôt disponible sur
GithubR, un environnement virtuelrenvvous permet de recréer la même configuration logicielle que celle utilisée pour générer cette page. Pour cela, il suffit de fairerenv::restore().↩︎Pour en savoir plus sur ce projet, se rendre sur la documentation du projet.↩︎
Ce filtre est construit après lecture du dictionnaire des variables de la BPE.↩︎